摘 要: 基于對無標記數據算法的研究,討論了基因數據分析的半監督學習算法。基因數據的典型特征是小樣本、高維數,處理起來非常困難。在安全的半監督學習基礎上,提出了一種降維和半監督學習相結合的方法,以提高分類效果的精確度及魯棒性。實驗證明,該方法通過結合降維和半監督學習的優點,具有很好的應用價值。
基因芯片技術可以一次性對大量基因序列進行檢測和分析,從而得到高維的基因表達數據,因此在病理研究和臨床應用等領域備受關注[1-2]。基因表達數據中基因的數目巨大,大部分都無法為樣本的區分提供有用信息,因此識別出一小部分能提供足夠有用信息的基因并實現很好的分類至關重要,這一小部分有效基因被稱為分類特征基因。特征基因選擇通常由去除分類無關基因和去除冗余基因兩部分組成。GOLUB T R等人提出的特征記分準則FSC(Feature Score Criterion)用于去除分類無關基因[3]。李穎新等人[4]認為應該在此基礎上考慮方差對樣本分類的影響,利用方差不同對樣本分類的貢獻不同,從而更客觀地評價基因包含的分類信息量,提出了修訂的特征記分準則RFSC(Revised Feature Score Criterion)。關于特征基因的選擇研究有過濾法、纏繞法、混合方法等[5-6]。特征基因選擇之后,選出的剩余基因可以看作與疾病相關。因為利用基因數據的高維數和高噪音進行特征提取是必要的途徑。本文結合降維技術方法給出新的特征提取方法。提取特征后,樣本的分類又顯得尤為重要,一個好的分類器能夠更準確、更有效地區分正常樣本,從而為臨床醫學提供參照。半監督學習(Semi-supervised Learning)是目前比較有效的分類方法[7],它主要考慮如何利用少量的標注樣本和大量的未標注樣本進行訓練和分類的問題。本文提出的降維和半監督學習方法能夠更好地利用數據的隱含信息,更好地實現分類預測效果。
1 數據描述及標準化
1.1 基因表達數據
基因表達譜數據可以表述為:
M=x11 x12 … x1n c1x21 x22 … x2n c2 ?塤xm1 xm2 … xmn cm (1)
腫瘤基因表達譜M中共有m個樣本、n個基因。xij代表第i個樣本的第j個基因表達值; ci代表樣本所屬類別。gi為第i個基因所有樣本的表達值,簡稱為基因gi:
gi=x1ix2ixmi (2)
基因表達譜最顯著特點是樣本少、維數高,即m<<n[2]。
1.2 基因表達數據標準化
對基因表達數據進行標準化的方法主要有兩種:(1) 限制每個基因的均值為0,方差為1;(2)限制每個基因的值在[0,1]范圍內。
本文采用限制每個基因的值在[0,1]范圍內的標準化方法:
xij*=(xij-xj-max-xj-min)
其中,xij為式(1)中所示的基因表達矩陣M中第i個樣本的第j個基因,xj-max為第j列基因的最大值,xj-min為第j列基因的最小值,xij*為標準化后的新值。
2 數據處理的相關理論方法
2.1 IRFSC特征計分準則
由于相關組織的某些基因發生了突變,而突變基因的表達水平與正常基因的表達水平不一樣,因此,需要找出突變基因。原則是:將患病和正常樣本兩類中具有顯著差異的基因子集保留,其余視為無關基因去除。通常是按照某種記分準則對每一個基因進行記分[6],分值越大說明該基因的正常樣本和腫瘤樣本差異越大、基因含有的分類信息就越多。因此按基因分值大小降序排列,排在前面一定數量的基因作為候選基因,其余基因作為無關基因去除。參考文獻[5]中對GOLUB T R等人提出的FSC進行改進后為:
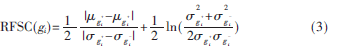
若基因表達譜中只有正常樣本和腫瘤樣本兩類,則表示基因gi中正常樣本的均值,表示gi中腫瘤樣本的均值;表示gi中正常樣本的標準差,表示gi中腫瘤樣本的標準差。考慮到樣本數目m和基因數目n之間的大小關系m<<n[2],由于n/m的比值越大,對方差的影響也越大,給出式(3)的改進形式:
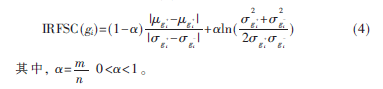
2.2 降維方法
2.2.1 主成分分析
主成分分析PCA(Principal Component Analysis)算法是一個經典的統計技術,把數據從高維的空間中映射到低維的空間并保留主要的信息[8]。在新的坐標系下,變換數據點的方差沿新的坐標軸得到了最大化,這些坐標軸經常被稱為主成分。PCA的主要運算步驟如下:

2.2.2 局部保持投影
局部保持投影LPP(Locality Preserving Projection)是一種線形的降維算法[9]。LPP的目的是保持局部的結構信息,所以在低維空間的近鄰搜索與高維空間產生類似的結果。LPP是線性的,這使得LPP處理速度很快,并且很適合于實際的應用,這也是LPP優于LLE的地方。降維后產生的變換矩陣可以直接應用在測試集上,所以,其擁有樣本外點學習能力。
LPP的算法如下:
(1)PCA投影。通過保留主要成分,將數據集投影到子空間。
(2)構建鄰近圖。如果樣本點Xi和樣本點Xj是近鄰點,則可以在Xi和Xj之間建立一條邊。建立的近鄰圖是局部流形結構的近似,常采用K近鄰方法。
(3)如果樣本點i和j相連,則設置權重W=e,其中t是一個合適的常數;否則W=0。
其中,L=D-W,而對角矩陣D的對角線元素Dii=Wij。
(4)本征映射。計算特征值分解問題:XLXT?琢=?姿XXT?琢求解廣義特征方程的d個最小特征值對應的特征向量作為d個投影向量。故由上述特征方程的d個最小特征值?姿1,?姿2,…,?姿d對應特征向量?琢1,?琢2,…,?琢d,構成保持近鄰重建特性的線性變換矩陣。
2.3 支持向量機
支持向量機SVM(Support Vector Machines)是由VAPNIK提出的一種新的機器學習方法[10],它基于VC維和結構風險最小化理論 (SRM),在很大程度上解決了傳統機器學習中的維數災難及局部極小等問題。由于其完整的理論框架和在實際應用中取得了很多好的效果,在機器學習領域受到了廣泛的重視,圖1給出了SVM分類的示意圖。

當給定的訓練樣本集為{xi,yi},其中i=1,…,N,相應的類標簽為yi={-1,+1}。在線性可分的情況下,SVM 能找到一個能使兩類樣本集分類間隔最大的最優超平面。這等價于解決下面的規劃問題:
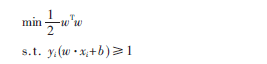
由于支持向量機是利用有效的少量樣本去構建超平面來預測測試樣本,支持向量的選擇對分類器的精度影響很大。對大量的數據樣本進行處理,分類器的精度變化不會很大;但是對于少量的樣本進行處理,分類器精度的變化會非常明顯,尤其是遇到高維小樣本問題。分類器的精度如果變化很大,就在實際應用中就會帶來意想不到的后果。鑒于基因數據的特點(高維數和高噪音),把最新的支持向量機S4VM應用到本文的算法中。S4VM是對S3VM的改進,后者主要關注一個最優的低密度分界線,而S4VM同時關注多個可能的低密度分界線。因為給定一些有標記的點和大量為標記的點,可能存在不止一個低密度分界線,所以很難決定哪個是最好的,如圖2所示。雖然這些低密度分界線都與有限的標記樣本吻合,但它們之間的差異很大,因此如果選錯了,會有一個很大的損失,導致性能的下降。所以,S4VM試圖考慮所有可能的低密度分界線。在給定許多不同“間隔”較大的分界線時,通過對未標記的樣本的類別劃分進行優化,使得在最壞的情況下,相對于只使用標記樣本的支持向量機的性能提升最大化。

3 算法流程
本文提出算法的具體流程如下。
(1)對腫瘤基因表達譜進行標準化。
(2)去除無關基因。利用RFSC計算每個基因的分值,
經過降序排列后選出分值靠前的一部分基因作為候選基因。
(3)特征提取。利用局部保持投影提取主要的特征。
(4)分類測試。利用S4VM進行分類精確度的檢驗。
4 實驗結果及討論
采用腫瘤基因表達數據集(http://home.ccr.cancer.gov/ oncology/oncogenomics/)進行實驗測試,該表達譜共27個樣本(其中16個為正常樣本,11個為腫瘤樣本),3 467個基因。然后,采用ALON等人[11]選出的含有2 000個特征基因的結腸癌基因表達譜數據集,包括40個結腸癌組織樣本和22個正常樣本進行降維實驗對比。
實驗1 選取數據集中的27個樣本,采用SVM和S4VM進行分類精度檢驗,分別選取分類信息指數為0.5、0.7、0.8、0.85、0.9的2 085、819、417、279和184個基因。SVM選取1/10、3/10、5/10、7/10、8/10作為訓練集;S4VM選取1/10、3/10、5/10、7/10、8/10作為有標號的數據。實驗對比結果如表1所示。
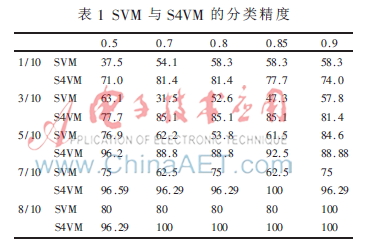
在此實驗中,訓練集的樣本數越多,分類效果越好;對于S4VM,有標記的樣本數越多,其精確度越高,而且可以看出S4VM不會出現太大的精度變化,而SVM精度變化很大,說明S4VM的魯棒性效果非常好。
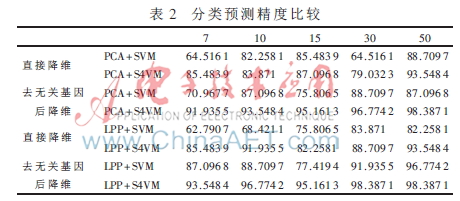
實驗2 進行去無關基因和直接降維的對比。采用了PCA+SVM、PCA+S4VM、LPP+SVM及LPP+S4VM進行了對比實驗;選擇315個特征基因分別降到7、10、15、30、和50維。采用SVM分類器時作為訓練集,在用S4VM時采用62個樣本中的3/10約 19個作為標記的數據。實驗對比結果如表2所示。可以得出,無關基因作為樣本中無關的屬性,其存在對分類器模型的選取影響很大;LPP由于很好地保持了局部結構,其降維效果明顯要比PCA好;S4VM由于考慮了多個低密度分割器,并且充分利用了無標記的數據,使其對分類器的訓練有更好的貢獻,其魯棒性及精度要比直接使用標記數據的SVM分類效果更明顯。
基因數據的主要特點是高維數和高噪音。本文基于挖掘數據本質特征的思路,采用降維和安全的機器學習技術提出一種基因數據分析的半監督學習算法。通過實驗證明了算法中應用降維的有效作用,同時也顯示出應用S4VM作為學習機的潛力,為該方面更深入的研究提供了重要理論和方法基礎。
參考文獻
[1] HOUBIN B, CHUNG R. Gene expression data classificationand Bioengineering(BIBE), IEEE 11th Inter-national Conference, 2011:66-71.
[2] YEUNG K Y, RUZZO W L. Principal component analysis for clustering gene expression data[J]. Bioinformatics, 2011,7(9):763-774.
[3] GOLUB T R, SLONIM D K, TAMAYO P, et al. Molecularclassification of cancer: class discovery and class predic-tion by gene expression monitoring[J]. Science, 1999(286):531-537.
[4] 李穎新,李建更,阮曉剛.腫瘤基因表達譜分類特征基因選取問題及分析方法研究[J].計算機學報,2006,29(2):324-330.
[5] 于化龍,顧國昌,趙靖,等. 基于DNA微陣列數據的癌癥分類問題研究進展[J]. 計算機科學,2010,37(10):16-22.
[6] 張敏,戈文航.雙聚類的研究與進展[J]. 微型機與應用,2012,31(4):4-6.
[7] HUANG S C, WU T K. Robust semi-super-vised SVM on kernel partial least discriminantspace for high dimensional data mining[C]. Proceedings of Information Science and Appli-cations(ICISA), International Conference,2012:1-6.
[8] JOLLIFFE T. Principal component analysis [M].New York:Springer,1986.
[9] He Xiaofei,NIYOGI P.Locality preserving pro-jections[C]. Proceedings of Advances In Neural Information Processing Systems 16, MA: Cambridge, MIT Press, 2004:153-160.
[10] Li Yufeng, Zhou Zhihua. Towards making unlabeled data never hurt[C]. In: Proceedings of the 28th International Conference on Machine Learning(ICML’11), Bellevue, WA, 2011:1081-1088.
[11] ALON U, BARKAL N, NOTTERMAN D A, et al. Broad patterns of gene expression revealed by clustering analysisof tumorand normal colon tissues by oligonucleotide arr-ays[J]. Proceedings of the National Academy of Science, 1999,96(12):6745-6750.

